培地成分を対象としたベイズ最適化の活用入門(バッチ最適化編)
培地成分を対象としたベイズ最適化の活用入門(バッチ最適化編)
■はじめに
前回の記事では、逐次最適化を用いて、ラウンドごとに1条件ずつ生成し、その都度モデルを更新していくアプローチを紹介しました。しかし実際の実験では、1ラウンドで複数の条件を並行して試すことが一般的です。そこで今回は、そのような「バッチ最適化」の実装例を紹介していきます。
今回も細胞シミュレーションのために、前回登場した「細胞くん」を対象に、最適な培地条件を探索します。振り返りとして、この「細胞くん」関数はグルコース量を x、窒素源 NH₄Cl を y として、ある挙動を示す2次元関数です。分布は以下のようになります。
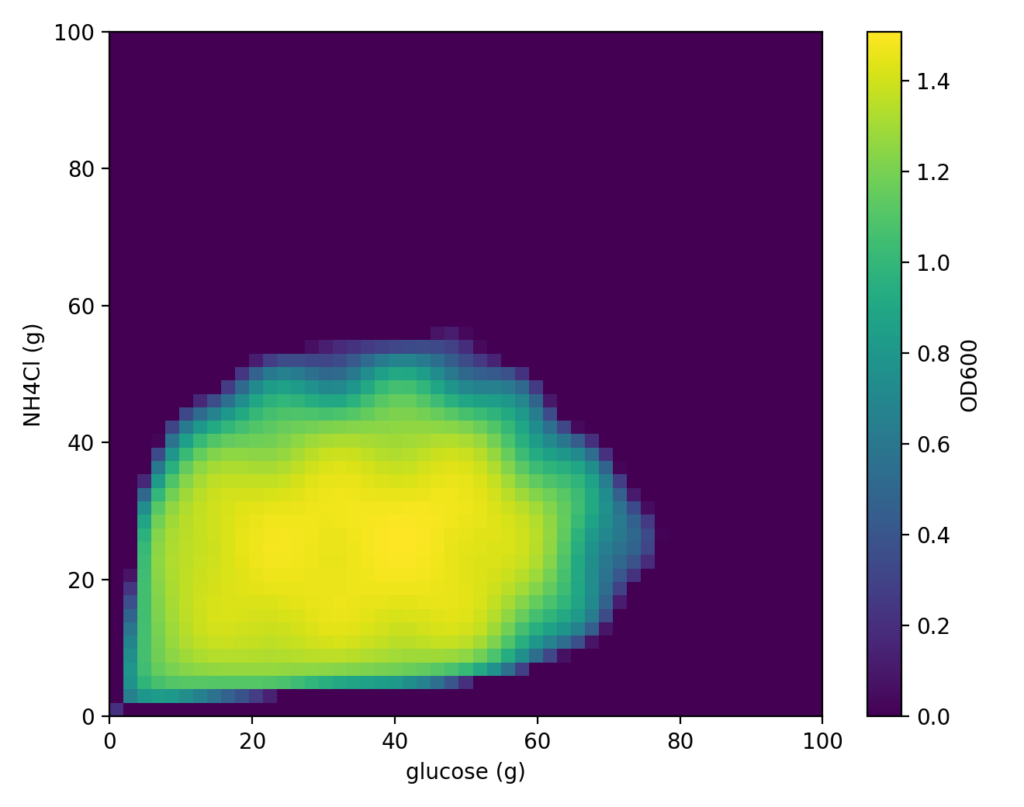
細胞くんのグルコースとNH₄Cl量に対する応答をシミュレーションした関数の分布
前回に引き続き、初期データとして、以下の実測値を設定します。
| Glucose (g) | NH4Cl (g) | OD600 |
|---|---|---|
| 5.0 | 5.0 | 0.761678 |
| 5.0 | 95.0 | 0.874593 |
| 95.0 | 5.0 | 0.879424 |
| 95.0 | 95.0 | 0.902424 |
| 50.0 | 50.0 | 1.079207 |
以降のスクリプトは、次のリンクから実行できます。
まず解析に必要なパッケージ群をインストールします。
!pip -q install imageio openpyxl scikit-optimize次に、初期データをPythonで利用可能なデータフレームに読み込みます。
import pandas as pd
from io import StringIO
csv_text = """Glucose (g),NH4Cl (g),0D600
5.0,5.0,1.079207
5.0,95.0,0.000000
95.0,5.0,0.000000
95.0,95.0,0.000000
50.0,50.0,0.510030
"""
# 文字列CSV → DataFrame
df_raw = pd.read_csv(StringIO(csv_text))
# 列名を標準化:x=glucose, y=NH4Cl, od=OD600
seed_df = df_raw.rename(columns={"Glucose (g)": "x", "NH4Cl (g)": "y", "0D600": "od"})
# 確認
seed_df続いて、ベイス最適化に用いるモデルを準備します。
from od_objective_2d import ODObjective2D, BOUNDS2D
from skopt import Optimizer
from skopt.space import Real
import numpy as np
# 目的関数(細胞くん)の生成
obj = ODObjective2D()
# 探索範囲(x=glucose, y=NH4Cl の下限/上限)を定義
space = [Real(BOUNDS2D["x"][0], BOUNDS2D["x"][1], name="x"),
Real(BOUNDS2D["y"][0], BOUNDS2D["y"][1], name="y")]
# ベイズ最適化器の生成
opt = Optimizer(
dimensions=space,
base_estimator="GP", # 内部の代理モデルにガウス過程 (Gaussian Process) を使用
acq_func="EI", # 獲得価値関数:改善期待値 (Expected Improvement) を使って「次に測るべき点」を決めます。
random_state=7
)
best_od = -np.inf
best_xy = (None, None)
rows = []
for i, r in seed_df.reset_index(drop=True).iterrows():
xi, yi, odi = float(r["x"]), float(r["y"]), float(r["od"])
opt.tell([xi, yi], -odi)
if odi > best_od:
best_od, best_xy = odi, (xi, yi)
rows.append({"iter": i+1, "phase": "seed", "x": xi, "y": yi, "od": odi,
"best_so_far": best_od, "best_x": best_xy[0], "best_y": best_xy[1]})
ここで
ODObjective2D は「細胞くん」の挙動を模擬する関数です。実際の実験ではこの部分をスキップし、実測値を代わりに入力します。
■バッチ最適化の実行
ここから前回の実装と変わっていきます。
n_rounds = 10 # ラウンド数(各ラウンドで batch_size 個の条件を提案)
batch_size = 2 # 1ラウンドで何条件提案するか
print("\n=== ベイズ最適化(バッチ提案)を開始 ===")
for r in range(1, n_rounds + 1):
# 1) ask: 次に測るべき点を batch_size 個まとめて提案
X_next = opt.ask(n_points=batch_size, strategy="cl_min")
print(f"[BO round {r}] 提案点:")
for j, (x_next, y_next) in enumerate(X_next, start=1):
print(f" cond{j}: Glucose={x_next:.3f}, NH4Cl={y_next:.3f}")
# 2) 実験(デモ: 目的関数で代用。実運用では実測ODに置換)
od_list = [float(obj(xn, yn)) for (xn, yn) in X_next]
for j, odm in enumerate(od_list, start=1):
print(f" → cond{j} 測定 OD= {odm:.6f}")
# 3) tell: まとめて BOモデル に渡す
opt.tell(X_next, [-od for od in od_list])
# 4) ベスト更新チェック & ログ追加
for j, ((xn, yn), odm) in enumerate(zip(X_next, od_list), start=1):
improved = ""
if odm > best_od:
best_od, best_xy = odm, (xn, yn)
improved = " ← ★ベスト更新!"
rows.append({
"iter": len(rows) + 1,
"phase": "bo",
"round": r,
"cond_in_round": j,
"x": xn,
"y": yn,
"od": odm,
"best_so_far": best_od,
"best_x": best_xy[0],
"best_y": best_xy[1],
})
print(f" cond{j}: 現在のbest = {best_od:.6f} @ {best_xy}{improved}")
# skopt 内部での最良点(-OD を最小化した結果)を確認
best_idx = int(np.argmin(opt.yi))
skopt_best_xy = tuple(opt.Xi[best_idx])
skopt_best_od = -opt.yi[best_idx]
print("\n=== 最終サマリ ===")
print(f"ログ上の best: OD={best_od:.6f} @ {best_xy}")
print(f"skopt の best: OD={skopt_best_od:.6f} @ {skopt_best_xy}")今回は以下の設定で最適化を実施しました。
n_rounds = 10 # ラウンド数(各ラウンドで batch_size 個の条件を提案)
batch_size = 2 # 1ラウンドで何条件提案するか各ラウンドで2条件ずつ提案し、それぞれを評価してモデルに反映させます。結果として得られた最適条件は以下の通りです。
=== ベイズ最適化(バッチ提案)を開始 ===
[BO round 1] 提案点:
cond1: Glucose=98.874, NH4Cl=85.862
cond2: Glucose=98.328, NH4Cl=10.249
→ cond1 測定 OD= 0.000000
→ cond2 測定 OD= 0.000000
cond1: 現在のbest = 1.079207 @ (5.0, 5.0)
cond2: 現在のbest = 1.079207 @ (5.0, 5.0)
[BO round 2] 提案点:
cond1: Glucose=57.559, NH4Cl=98.794
cond2: Glucose=64.765, NH4Cl=55.086
→ cond1 測定 OD= 0.000000
→ cond2 測定 OD= 0.000000
cond1: 現在のbest = 1.079207 @ (5.0, 5.0)
cond2: 現在のbest = 1.079207 @ (5.0, 5.0)
[BO round 3] 提案点:
cond1: Glucose=35.263, NH4Cl=53.854
cond2: Glucose=24.801, NH4Cl=9.849
→ cond1 測定 OD= 0.266920
→ cond2 測定 OD= 1.370388
cond1: 現在のbest = 1.079207 @ (5.0, 5.0)
cond2: 現在のbest = 1.370388 @ (24.800715628900235, 9.848637049589822) ← ★ベスト更新!
~中略~
[BO round 10] 提案点:
cond1: Glucose=29.950, NH4Cl=29.855
cond2: Glucose=50.098, NH4Cl=33.738
→ cond1 測定 OD= 1.472082
→ cond2 測定 OD= 1.455068
cond1: 現在のbest = 1.472082 @ (29.949798427708167, 29.85455402401584) ← ★ベスト更新!
cond2: 現在のbest = 1.472082 @ (29.949798427708167, 29.85455402401584)
バッチ最適化の様子を以下の折れ線グラフにまとめてみました。
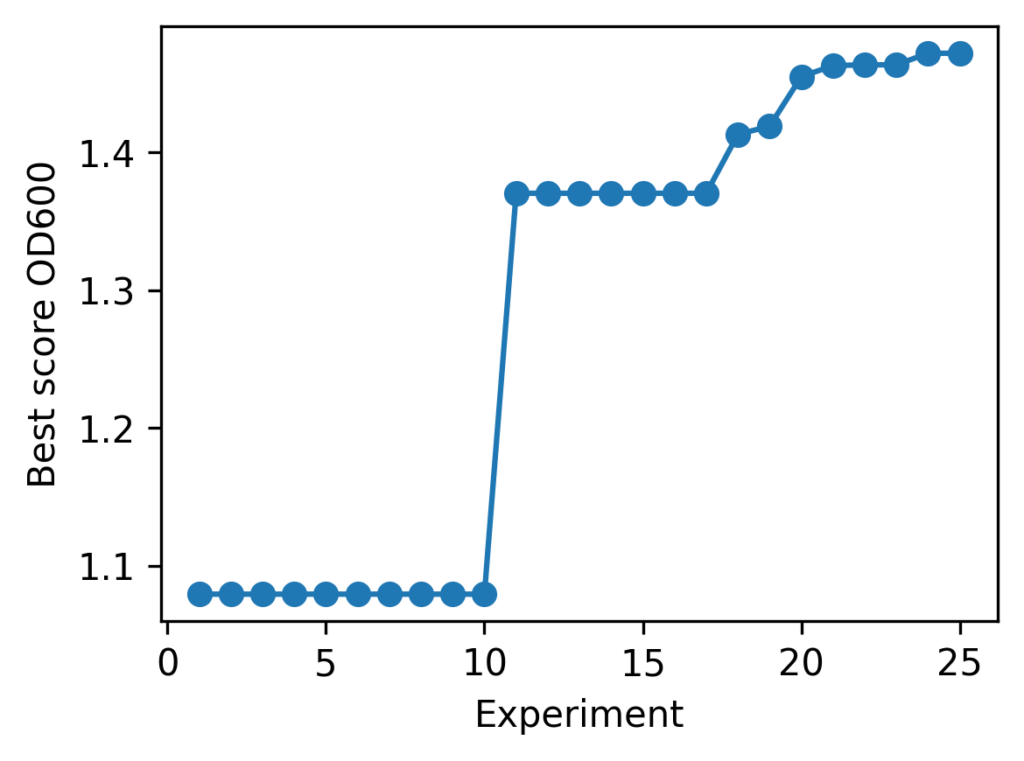
結果としては、以下の通りの培地組成が最適解として探索されました。
| best_od | best_glucose (g) | best_NH4Cl (g) |
|---|---|---|
| 1.472082 | 29.949798427708167 | 29.85455402401584 |
■シングルバッチ最適化との比較
ここまで、バッチ最適化の実装例と最適化結果を紹介してきました。 では、1条件ずつモデルを更新するシングルバッチ最適化とバッチ最適化はどのように違うのでしょうか。
同じ「細胞くん」を対象に最適化を行った結果を比べると、その差が明確になります。以下は、前回のシングルバッチ最適化(Sequential optimization)と今回のバッチ最適化(Batch optimization)の結果です。
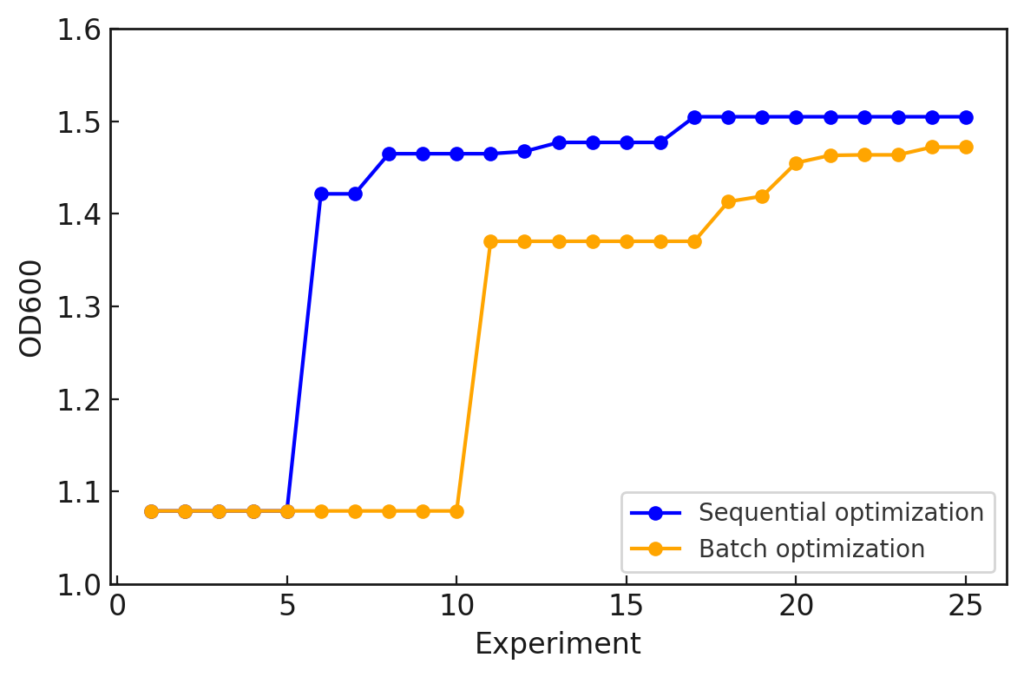
シングルバッチ最適化の方が探索が早く収束し、性能もやや高いことが分かります。シングルバッチ最適化では1実験ごとにモデルを更新できるため、一般的に探索効率が高いです。一方で、バッチ最適化は並行実験が可能ですが、その分モデルの更新頻度が少なくなるため、探索効率は下がります。
ただし、現実の研究では「1回の培養に1ヶ月かかる」といった状況もあり、その場合は1バッチでの逐次最適化は非現実的です。実験効率を考慮すると、バッチ最適化が実務的には有用になります。つまり、探索精度と実験効率のトレードオフが存在します。
研究環境に応じてどちらを優先するべきかが決まります。今回は、バッチ最適化の実装例とシングルバッチ最適化との比較を通じて、その特徴を紹介しました。本記事の細胞挙動を模倣した一連のシミュレーションで、ライフサイエンス分野での応用イメージを持っていただければ幸いです。
実行環境について
本ページ記載のプログラムは Google Colab を用いて動作確認しています。
再現性を確保するため、Python のバージョンおよび主要ライブラリのバージョンを以下に記載します。
最終動作確認日:2025年11月10日
Python バージョン
Python 3.10.12 (Google Colab デフォルト)
使用ライブラリのバージョン
| ライブラリ | バージョン |
|---|---|
| dataclasses | 0.6 |
| imageio | 2.37.0 |
| matplotlib | 3.10.0 |
| numpy | 2.0.2 |
| openpyxl | 3.1.5 |
| pandas | 2.2.2 |
| scikit-optimize | 0.10.2 |
